如何快速高效进行突变体检测和鉴定是植物基因组编辑技术迅速发展面临的重要问题之一。目前植物基因组编辑突变检测方法主要包括PCR/RE、T7EI错配切割、临界退火温度PCR (ACT-PCR)、Sanger测序和二代测序(NGS)等。以上所有的检测方法都基于PCR反应,且都有各自的不足之处。PCR/RE方法的需要设计含有限制性内切酶位点的靶位点;T7EI无法区分纯合突变体和野生型以及杂合突变体与双等位突变体;ACT-PCR对PCR反应条件要求极高,而且无法检测到杂合突变;Sanger测序和NGS的价格比较昂贵,尤其是对于数目比较大的群体。
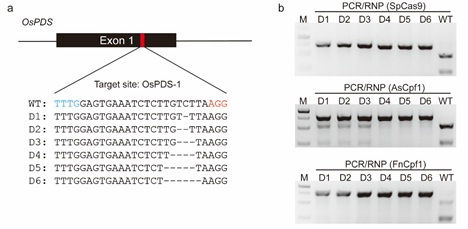
针对上述问题,中国科学院遗传与发育生物学研究所高彩霞研究组利用CRISPR/Cas系统(包括Cas9和Cpf1)的体外切割特性,在六倍体小麦和二倍体水稻中建立了一种简单、高效、廉价的PCR/RNP植物突变体筛选策略。该方法不受限制性内切酶位点的限制,比PCR/RE具有更强的广适性;比T7EI具有更高的准确度;比Sanger测序更廉价,而且灵敏度更高。基于SpCas9和FnCpf1 RNPs的PCR/RNP方法可以用于检测基因组编辑中经NHEJ修复产生的所有indel突变;基于FnCpf1 RNPs的PCR/RNP方法可以用于检测位于种子区域(seed region)内的SNP突变。该方法尤其适用于小麦的瞬时表达基因组编辑体系,如本实验室之前建立的CRISPR/Cas9 IVTs和RNPs介导的DNA-free基因组编辑体系。这是由于瞬时表达基因组编辑体系在后续组织培养过程中不使用任何的筛选标记,在T0代会获得较多的待筛选植株,而且PCR/RNP方法可以使用通用引物对发生在小麦A组、B组和D组各拷贝的突变进行同时检测,不会受靶位点周围SNPs的影响。此外,PCR/RNP方法也可以用于检测TALEN蛋白所诱导的突变。
该研究成果于2018年5月3日在线发表于Plant Biotechnology Journal杂志上(DOI: 10.1111/pbi.12938)。高彩霞研究组博士生梁振为该论文的第一作者。相关研究得到科技部、北京市科委、中科院、以及国家自然基金委的资助。