基因组测序及解析以及新技术的广泛应用,让人们继续探索着丝粒和端粒等染色体上高度重复区域在生命活动中的新功能。植物着丝粒含有丰富的重复序列,如串联重复序列(Satellite)和反转座子(Retrotransposon),参与基因组空间构象和细胞分裂等重要的生物学功能。然而不同物种双着丝粒染色体和新着丝粒染色体的不断发现,说明着丝粒区域大量的重复序列既不是其功能的充分条件也不是必要条件(Birchler and Han 2018; Liu et al., 2015)。因此我们一直想知道着丝粒这些特异的重复序列在着丝粒功能和结构维持中发挥的作用以及着丝粒在失去活性和产生活性等过程的遗传机制。
中国科学院遗传与发育生物学研究所韩方普研究组利用玉米为材料,利用着丝粒特异表观标记CENH3-RIP技术建立玉米着丝粒RNA文库并通过高通量测序以及克隆筛选等方法,意外发现来自玉米着丝粒特异反转座子序列CRM1通过反式剪切(Back splicing)的方式产生环RNA(图1)。研究人员进一步通过分子生物学(Divergent primers PCR、Northern等)、基因组学和生物影像观察(原子力显微镜)等方法证实重复序列来源的环状RNA位于着丝粒区域。功能研究发现CRM1来源的环RNA通过R-loop结构结合在着丝粒区域,并影响着丝粒区域高级染色质结构,遗传学的结果显示环RNA水平的降低能够影响CENH3核小体的定位。这些结果说明重复序列在着丝粒空间构象的维持和功能的发挥中发挥重要的作用(图2)。
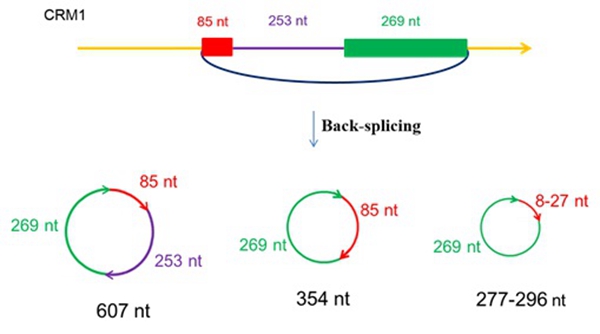
图1: 玉米着丝粒反转座子CRM1产生的环形RNA
韩方普研究组通过植物原生质体瞬时转化进行反式剪切体外实验,证实反转座子序列发生的反式剪切过程在含有大量反转座子的植物中发现(玉米,小麦,燕麦,高粱和大豆),然而在拟南芥和哺乳动物细胞系中没有发现。小麦非编码RNA数据也证实重复序列来源的环RNA在体内也真实存在,这些结果说明反转座子反式剪切的过程非常保守。植物尤其是玉米、小麦等作物的基因组包含大量的反转座子序列,这为后续研究重复序列在植物基因组进化以及染色质结构中的功能提供了一个新的角度。
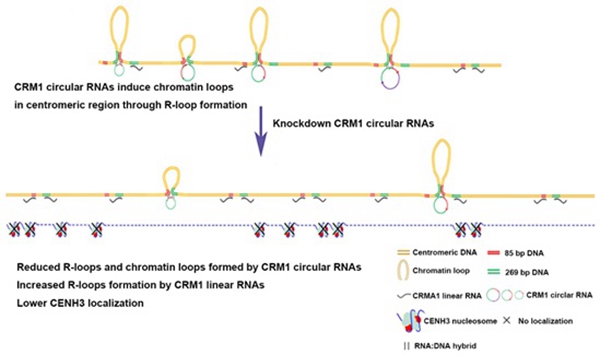
图2 : CRM1产生的环RNA在着丝粒空间构象和功能维持中作用
该论文于2020年1月29日在线发表于PLOS Biology(DO1:10.1371/journal.pbio.3000582),韩方普研究组已毕业博士研究生刘亚林和苏汉东为该文章的共同第一作者,韩方普研究员为通讯作者。本项工作得到遗传发育所曹晓风实验室、生物物理所李国红和俞洋实验室在RNA功能分析的帮助,遗传发育所黄勋和丁梅实验室在动物细胞系验证RNA剪切,物理所李伟实验室帮助完成原子力显微镜观察环RNA的实验。该研究得到国家自然科学基金重点国际合作项目的资助。