地球上所有的食物均直接或间接地来自于植物光合作用(Photosynthesis)。核酮糖-1,5-二磷酸羧化/加氧酶(Ribulose-1,5-bisphosphate carboxylase/oxygenase, Rubisco)是光合作用碳反应的关键酶。自然界存在最多的Rubisco由八个叶绿体基因组编码的大亚基(Rubisco large subunit, RbcL)和八个核基因组编码的小亚基(Rubisco small subunit, RbcS)组成。Rubisco的催化效率对提升农作物的产量起着决定性的作用。但是,由于目前对Rubisco的生物合成途径尚不清楚,阻碍了人们利用遗传学的手段使其在体外或异源表达。 Rubisco的生物合成包括折叠和组装两个部分,RbcL的折叠依赖分子伴侣素,而RbcL和RbcS的组装依赖组装因子。植物细胞与染色体工程国家重点实验室刘翠敏研究组长期从事Rubisco生物合成、结构与功能研究。研究组围绕Rubisco生物合成分子伴侣素Cpn60的结构与功能开展研究,取得了一系列进展(Nature Plants (2021), Plant J (2019), BMC Biology (2016), Mol Plant (2016))。近期,刘翠敏研究组在体外,利用真核生物拟南芥(Arabidopsis thaliana)的组装因子完成原核生物Synechococcus elongatus PCC6301 Rubisco的折叠与组装,产生有酶活的Rubisco。发现Syn6301 Rubisco accumulation factor 1 (Syn6301Raf1)具有真核组装因子Arabidopsis thaliana Rubisco accumulation factor 1 (AtRaf1)和Arabidopsis thaliana Bundle Sheath Defective 2 (AtBsd2)两者的功能,且经分子伴侣素GroEL/GroES折叠的RbcL必需真核组装因子AtRaf1和AtBsd2才能组装形成全酶,AtBsd2作用于AtRaf1的下游。冷冻电镜结构RbcL8,RbcL-AtRaf1和RbcL-AtRaf1-AtBsd2表明,Syn6301Raf1α结构域与RbcL的结合力强于AtRaf1α结构域与RbcL的结合力,这使得柔性较强的AtRaf1β结构域无法发挥其稳定RbcL8核心的功能,因此必需Bsd2帮助稳定Rubisco的柔性区域,从而完成Rubisco的组装。该研究进一步解析了原核与真核Rubisco生物合成途径。
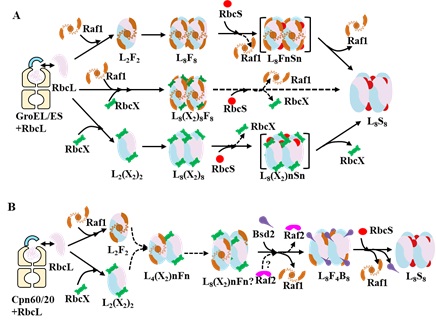
A图:原核Rubisco生物合成途径;B图:真核Rubisco生物合成途径
该研究成果以“Structural insight into the functions of Raf1 and Bsd2 in hexadecameric Rubisco assembly”为题于2023年10月18日在线发表于Molecular Plant(DOI:10.1016/j.molp.2023.10.011),刘翠敏研究员为通讯作者。刘翠敏研究组博士研究生王冉、宋辉博士为该文章的共同第一作者。该研究得到中国科学院战略性先导科技专项、国家重点研发计划和国家自然科学基金的资助。